重磅!PCAWG联盟6篇Nature、15篇Nature子刊以前所未有的规模揭示癌症的复杂性 |
 |
2020年2月6日讯/100医药网BIOON/---一项大规模的国际努力对来自38个人体组织的2600多个肿瘤基因组进行了多方面研究,从而对癌症的遗传基础产生了丰富的见解。
早在2001年对第一个人类基因组进行测序后,肿瘤的全面基因组表征就成为癌症研究人员的一个主要目标。从那时起,测序技术和分析工具取得的进展使得这个研究领域蓬勃发展。在发表在最新一期Nature期刊的6篇论文中,全基因组泛癌分析(Pan-Cancer Analysis of Whole Genomes, PCAWG)联盟进行了迄今为止最全面、最雄心勃勃的癌症基因组荟萃分析。与以前的研究工作主要集中于癌症基因组的蛋白编码区域不同,PCAWG联盟分析了整个基因组。在6篇Nature论文中,每篇论文都仔细研究了癌症遗传学的一个重要方面---总之,他们的发现对于理解癌症的完整遗传复杂性至关重要。
在讨论这些分析的影响之前,至关重要的是要强调支持PCAWG联盟努力的大量数据和复杂的组织框架。该项目涉及一个来自4大洲的744个附属机构的跨学科科学家小组,这些科学家必须克服主要的技术、法律和伦理挑战,才能在保护患者数据的同时进行分布式分析。研究人员分为16个工作组,每个工作组专注于癌症基因组学的不同方面---比如,评估突变的复发或推断肿瘤的发生。
总体来说,PCAWG联盟总共对38种肿瘤类型进行了综合分析。这些研究人员对2658个全癌基因组进行了测序,同时对来自同一癌症患者的相匹配的非癌细胞基因组进行测序。这些数据得到1188个转录组---肿瘤中RNA转录本的序列和丰度---的补充。
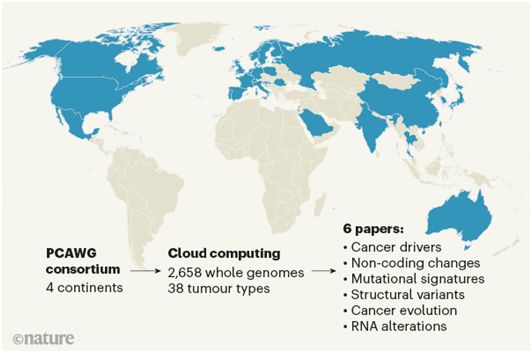 图片来自Nature, 2020, doi:10.1038/d41586-020-00213-2。
这些研究工作涉及广泛的质量控制和协调的数据处理,以及对用于检测突变的计算流程的大规模系统性实验验证。使用了许多计算算法和流程,并进行了比较。这需要数百TB的数据,这些数据分布在多个数据中心中,并且很可能需要数百万个处理小时---所有这些处理都由云计算实现。值得注意的是,PCAWG联盟的努力为云计算如何使得国际合作成为可能并有助于推进数据密集型领域发展提供了一个很好的例子。
图片来自Nature, 2020, doi:10.1038/d41586-020-00213-2。
这些研究工作涉及广泛的质量控制和协调的数据处理,以及对用于检测突变的计算流程的大规模系统性实验验证。使用了许多计算算法和流程,并进行了比较。这需要数百TB的数据,这些数据分布在多个数据中心中,并且很可能需要数百万个处理小时---所有这些处理都由云计算实现。值得注意的是,PCAWG联盟的努力为云计算如何使得国际合作成为可能并有助于推进数据密集型领域发展提供了一个很好的例子。
第一篇Nature论文概述了PCAWG数据集的广度和深度。PCAWG联盟报道平均而言,每个癌症基因组均携带4或5个驱动突变,这就为癌细胞提供了选择性优势。在研究的肿瘤中,仅5%没有发现驱动突变。相比之下,许多癌症表现出基因组灾难---复杂染色体重排(chromoplexy, 存在于17.8%的肿瘤)和染色体碎裂(chromothripsis , 存在于22.3%的肿瘤)---的特征,从而导致基因组发生重大的结构变化。
其他5篇Nature论文更详细地探讨了这个数据集的不同方面。比如,在第二篇Nature论文中,Rheinbay等人着手鉴定出非编码DNA中的遗传驱动因子。这是一项雄心勃勃的工作,这是因为要准确地检测非编码区中的突变比编码区中的突变要困难得多,更不用说评估非编码区中的突变。这些作者使用仔细的建模来排除伪影(artefacts),并系统地识别非编码驱动突变。
他们的结果使得以前报道的非编码驱动因子(比如长链非编码RNA NEAT1和MALAT1)受到质疑,但也揭示了新的非编码驱动因子。比如,这些作者报道了至关重要的肿瘤抑制基因TP53非编码区中发生的复发突变。他们还发现端粒酶基因TERT的非编码区中相对频繁发生的突变导致端粒酶的过度表达(这有助于肿瘤细胞不受控制地分裂),这与以前的一项针对晚期(转移性)肿瘤的泛癌研究中发现的较高端粒酶突变发生率(12%)相一致。尽管该研究不能排除存在其他非编码驱动因子,但它明确地表明这种类型的突变并不常见。
在第三篇和第四篇Nature论文中,Alexandrov等人和Li等人着重研究了称为标签(signature,,也译为签名,指的是独特性的DNA序列或单核苷酸位点)的基因组畸变(genomic aberrations)。不同的过程,比如缺陷性的DNA修复机制,或者暴露于环境诱变剂,会产生特征性的DNA畸变模式。如果我们要完善已知的突变特征并发现新的突变特征,那么更大规模的基因组数据集至关重要。令人印象深刻的是,Alexandrov等人和Li等人鉴定出97个标签序列。他们对先前研究工作的扩展不仅包括常规的单核苷酸标签,而且还包括涉及多核苷酸变异(multi-nucleotide variant)、小片段DNA插入或缺失的标签。
值得注意的是,Li及其同事们是最早发现涉及结构变异(structural variant)---基因组的大部分发生重排---的可重现标签的人之一。由于结构变异的多样性和复杂性,识别结构变异的过程比识别突变标签的过程更为复杂。
通过一系列的突变分组(mutation-subgrouping)步骤,这些研究人员确定了16个结构变异标签,比如揭示了两个结构变异标签---缺失和相互倒位(reciprocal inversion)---之间存在推定的机理联系。他们还了解所有16个标签在癌症中的作用。某些DNA修复基因中的突变显示与特征性的癌症标签相关。比如,PCAWG联盟发现基因CDK12中的突变与重复DNA的串联片段相关,并且DNA修复酶MBD4的截短变异与涉及称为CpG位点的DNA序列的独特突变标签同时出现。总而言之,这些新的标签为理解癌症产生机制以及诱变暴露在这个过程中的作用奠定了基础。
医药网新闻
- 相关报道
-
- JAMA:管住嘴、迈开腿,脑更灵!临床3期试验表明,坚持两年改善生活方式可显著改善老年人认知,加以监督、提高强度获益更多 (2025-08-01)
- 艾伯维2025H1:自免双子星116亿美元,FRα ADC增长76% (2025-08-01)
- 鹏瑞利集团、陆家嘴管理局和陆家嘴集团签订三方合作战略协议 拟设立上海首家外商独资三级综合医院 (2025-07-31)
- 为什么女朋友/老婆总爱改主意或纠结?最新Sci Adv:大脑中LINC00473对女性影响显著,使其敏锐地权衡沉没成本和风险收益 (2025-07-31)
- PLoS Comput Biol:人体生物钟在较高温下通过扭曲基因活动来保持24小时的节律 (2025-07-31)
- 喝含糖饮料竟会“改写”肠道细菌 DNA?Nat Commun:软饮料会影响肠道细菌和免疫系统之间的交流 (2025-07-31)
- Cell:指出一篇与冠状病毒抗病毒药物开发相关的研究存在缺陷 (2025-07-30)
- 《科学》:糖尿病治疗重大突破!科学家开发新型分子胶,可将RAS与PI3K亲和力提高500倍,无需胰岛素也可降糖 (2025-07-30)
- 打破昼夜节律局限!Nat Neurosci:CLOCK 基因揭秘人类大脑认知超凡进化的关键密码 (2025-07-30)
- ACS Nano:浙江大学研究发现,人类卵泡液和精浆中存在纳米塑料,影响受精能力和精子质量 (2025-07-30)
- 视频新闻
-
- 图片新闻
-
医药网免责声明:
- 本公司对医药网上刊登之所有信息不声明或保证其内容之正确性或可靠性;您于此接受并承认信赖任何信息所生之风险应自行承担。本公司,有权但无此义务,改善或更正所刊登信息任何部分之错误或疏失。
- 凡本网注明"来源:XXX(非医药网)"的作品,均转载自其它媒体,转载目的在于传递更多信息,并不代表本网赞同其观点和对其真实性负责。本网转载其他媒体之稿件,意在为公众提供免费服务。如稿件版权单位或个人不想在本网发布,可与本网联系,本网视情况可立即将其撤除。联系QQ:896150040






















