Nature Methods:能否通过数学模型揭示细胞周期的真实速度?VeloCycle有答案 |
 |

VeloCycle的原理与创新
VeloCycle的核心创新在于将基因表达状态映射到低维的流形上,再在流形上进行速度场的学习。流形(Manifold)可以理解为一个低维的几何空间,能够描述细胞基因表达状态的复杂变化。通过将RNA速度估计与低维流形相结合,VeloCycle可以保证速度向量在几何上保持一致,从而提高模型的准确性。
在数学上,VeloCycle基于贝叶斯生成模型,将细胞在基因表达空间中的运动描述为流形上的点的变化。研究人员通过随机变分推断(Stochastic Variational Inference, SVI)对模型的参数进行估计,并且利用了Pyro这一概率编程语言来实现这些推断过程。在对基因的速度参数(例如剪接速率 和降解速率 )进行估计时,VeloCycle可以将这些参数与低维流形上的坐标联系起来,从而实现统一的动力学推断。
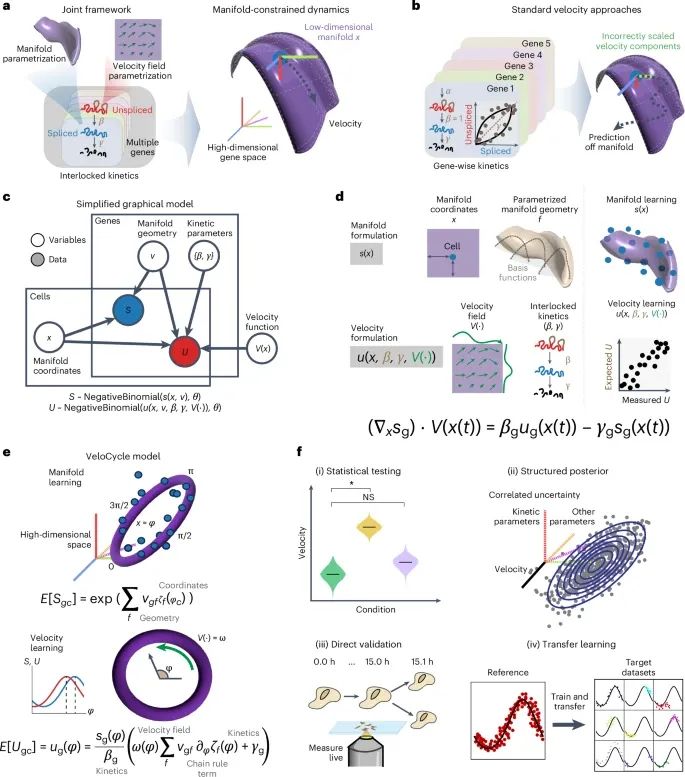
基于流形约束的RNA速度统计推断框架用于细胞周期研究(Credit:Nature Methods)
VeloCycle方法的整体框架和不同步骤
a:联合框架的示意图,展示了基因表达流形(manifold)和RNA速度场(velocity field)的参数化联合框架。流形描述了基因表达在低维空间中的变化,而速度场则描述了基因表达变化的动态。
b:传统方法的无约束速度估计,对比展示了传统RNA速度推断方法的示意图,这些方法往往缺乏对速度方向的几何约束,导致速度估计在不同基因之间不一致。
c:概率关系的图模型,用板图(plate diagram)展示了潜在变量(如基因表达状态S和未拼接RNA状态U)与可观测数据之间的概率关系:S是基于期望值、流形坐标和流形几何参数采样得到的。U是基于流形信息、动力学参数(例如 和 )以及速度函数采样得到的。坐标定义了每个细胞在潜在空间中的位置,几何形态定义了沿着流形的表达变化。
d:流形与速度公式化,上半部分描述了对于剪接转录本(spliced counts, s)的流形公式化,使用细胞特定的坐标(x)和基因特定的几何族(f)来直接映射观测数据到高维空间。下半部分描述了未剪接转录本(unspliced counts, u)的速度公式化,通过速度场函数(V)和相关的动力学参数( , )得到,最终通过对这些实体的求导(应用链式法则),将速度描述为流形坐标x(t)的直接函数。
e:周期性过程中的流形约束速度估计,描述了VeloCycle如何在周期性生物过程中的应用,首先通过流形学习估计坐标和几何,接着通过速度学习估计动力学参数和速度函数。
f:VeloCycle的新型速度分析类型,展示了VeloCycle可以实现的新型分析:统计显著性测试:在多个样本之间以及对照零假设进行的统计显著性测试。后验边际分布分析:通过马尔科夫链蒙特卡罗(MCMC)采样对模型参数进行后验分布分析。速度外推到真实生物时间:使用活细胞显微镜数据验证。从大参考数据集到小目标数据集的迁移学习:可以将基因流形从大型参考数据集迁移到较小的数据集。
VeloCycle的模拟数据与模型验证
为了验证VeloCycle的有效性,研究人员使用了模拟数据和真实的单细胞RNA测序数据进行对比。模拟数据的设计旨在保持基因之间的动力学参数关系,这些关系在真实数据中是被预期的。例如,模拟数据中包含3000个细胞和300个基因的20个数据集,VeloCycle在这些数据集上推断的细胞周期阶段与真实值的圆形相关系数为0.95,表明了模型的高准确性。
此外,研究人员还进行了敏感性分析,测试了VeloCycle在不同数据集规模上的表现。当细胞数量为100个或基因数量为100个时,VeloCycle仍然能够保持较高的推断准确性,其圆形相关系数大于0.70。这表明,即使在较小的数据集下,VeloCycle也能够稳定地进行细胞周期速度的估计。
在与DeepCycle这一基于自动编码器的方法的对比中,VeloCycle在多次模拟中的平均均方误差(MSE)比DeepCycle低60%,且相关系数r为0.95,而DeepCycle的r值为0.73。这意味着VeloCycle在速度推断方面显著优于DeepCycle。
对细胞周期速度的估计与生物学验证
VeloCycle的另一个重要应用是在细胞周期速度的估计上。在对人类视网膜色素上皮细胞(RPE1细胞)进行研究时,VeloCycle推断的细胞周期持续时间约为17.7小时,这与通过时间延迟显微镜对同一细胞的实验测量值(17.7小时,标准差为3.4小时)非常接近。这一结果表明,VeloCycle在对细胞周期速度进行推断时,与实际的实验数据有很好的吻合度。
研究人员还在小鼠胚胎(mES细胞)上测试了VeloCycle,这是一种快速循环的细胞类型。VeloCycle推断出其平均细胞周期为10.5小时,这一估计与快速循环细胞的生物学特征高度一致。
敏感性分析与模型的鲁棒性
为了确保VeloCycle模型在不同情境下的可靠性,研究人员对其进行了全面的敏感性分析。例如,在真实速度值变化的情况下,VeloCycle仍然能够保持稳定的推断性能,且估计结果与真实值的偏差在0.2%到35.8%之间。对于速度较慢的情况,推断误差有所增加,这可能是由于较短的未拼接和拼接RNA之间的延迟难以准确刻画。然而,在所有模拟数据中,VeloCycle对于剪接和降解速率的比值的估计几乎完全匹配真实值,其平均相关系数为0.99。
此外,研究人员还验证了数据集的规模对模型的影响。结果表明,数据集中的细胞数量和基因数量对模型的准确性有显著影响。使用更多的细胞可以弥补基因数量的不足,反之亦然。研究人员建议,对于准确的速度估计,至少需要500个细胞和50个基因,或350个基因和50个细胞。
VeloCycle的强大之处在于其不仅能够用于模拟数据,还可以应用于各种真实的生物数据集。例如,研究人员将VeloCycle应用于小鼠胚胎干细胞和人类成纤维细胞的数据,这些数据来自不同的单细胞RNA测序技术和不同的生物样本。在所有这些数据集中,VeloCycle都能够成功地推断出细胞的周期状态,并且与现有的基于标记基因的分类结果高度一致。
在细胞周期速度的研究中,VeloCycle还展示了其在大规模基因敲除实验中的应用潜力。研究人员利用Perturb-seq数据集进行了实验,其中数百个基因敲除被引入RPE1细胞系。通过VeloCycle的推断,发现了一些特定基因敲除条件下细胞周期速度的显著变化,表明这些基因对细胞周期的进程具有重要影响。
VeloCycle为我们提供了一种全新的工具来研究细胞周期中的动态变化,通过结合低维流形学习与速度学习,它克服了传统RNA速度模型在统计和几何上的不一致性,为细胞周期的动力学推断提供了更加精确的手段。
医药网新闻
- 相关报道
-
- Bio-Protocol:北京妇产医院刘朝晖团队成功构建“内膜外翻”的人子宫内膜类器官(含详细操作步骤) (2025-09-18)
- 18款1类新药首次在中国获批临床! (2025-09-18)
- Cell:mRNA疫苗,进军肺结核领域,增加并超越卡介苗保护效果 (2025-09-18)
- Nature子刊:AI从头设计多肽,精准抑制细胞焦亡,开辟炎症疾病治疗新途径 (2025-09-18)
- Nature:胃的“南北战争”!神经组织竟是调控胃功能分区的幕后指挥官 (2025-09-18)
- Nature Medicine:胖,但健康!遗传学如何“解绑”肥胖与心脏病的宿命关联? (2025-09-17)
- 国内唯一获批干细胞药物首位患者出院,国产干细胞药品取得临床应用突破 (2025-09-16)
- 《自然》:慢性压力毁血糖!科学家发现全新调控血糖神经通路,可快速调节肝脏糖生成,反复压力会钝化此通路,导致糖代谢受损 (2025-09-16)
- FDA监管趋严,如何影响中国创新药出海? (2025-09-16)
- JAMA子刊:重大突破!北京安定医院团队首次证实,ANK3可以预测liafensine治疗难治性抑郁症的疗效 (2025-09-16)
- 视频新闻
-
- 图片新闻
-
医药网免责声明:
- 本公司对医药网上刊登之所有信息不声明或保证其内容之正确性或可靠性;您于此接受并承认信赖任何信息所生之风险应自行承担。本公司,有权但无此义务,改善或更正所刊登信息任何部分之错误或疏失。
- 凡本网注明"来源:XXX(非医药网)"的作品,均转载自其它媒体,转载目的在于传递更多信息,并不代表本网赞同其观点和对其真实性负责。本网转载其他媒体之稿件,意在为公众提供免费服务。如稿件版权单位或个人不想在本网发布,可与本网联系,本网视情况可立即将其撤除。联系QQ:896150040





















