Cell:中山大学联合阿里云,利用人工智能揭开隐藏的RNA病毒世界,一次性发现16万种新病毒 |
 |
来源:生物世界 2024-10-12 09:26
该研究揭示了有史以来最大的RNA病毒,其基因组长达47250个核苷酸,全方位刷新了人们对RNA“病毒圈”的认知。中山大学医学院施莽教授团队和阿里云李兆融团队合作,在国际顶尖学术期刊 Cell 上发表了题为:Using artificial intelligence to document the hidden RNA virosphere 的研究论文。
研究团队利用人工智能(AI)技术发现了180个病毒超群和16万余种全新RNA病毒,将已知病毒种类扩充了近30倍。其中包括传统研究方法未能发现的病毒 暗物质 ,极大扩展了全球RNA病毒的多样性。这一突破标志着深度学习算法在病毒发现领域取得了里程碑式的进展,为病毒学研究开创了一种全新的范式。

得益于深度学习在病毒发现领域及整个生物学背景下的广泛应用,并取得了初步进展,本研究前所未有地整合了序列信息与结构信息,运用深度学习方法对全球各地10487份宏转录组进行病毒挖掘,成功发现了513134条病毒基因组,代表161979个潜在病毒种类及180个RNA病毒超群(相当于门或纲的分类级别),使RNA病毒超群数量扩容约9倍。其中23个超群无法通过序列同源性方法识别,被称为病毒 暗物质 。
这些神秘的病毒存在于地球上每一种生境中,如空气、南极底泥、深海热泉、活性污泥和盐碱滩等。值得注意的是,该研究揭示了有史以来最大的RNA病毒,其基因组长达47250个核苷酸,全方位刷新了人们对RNA 病毒圈 的认知。
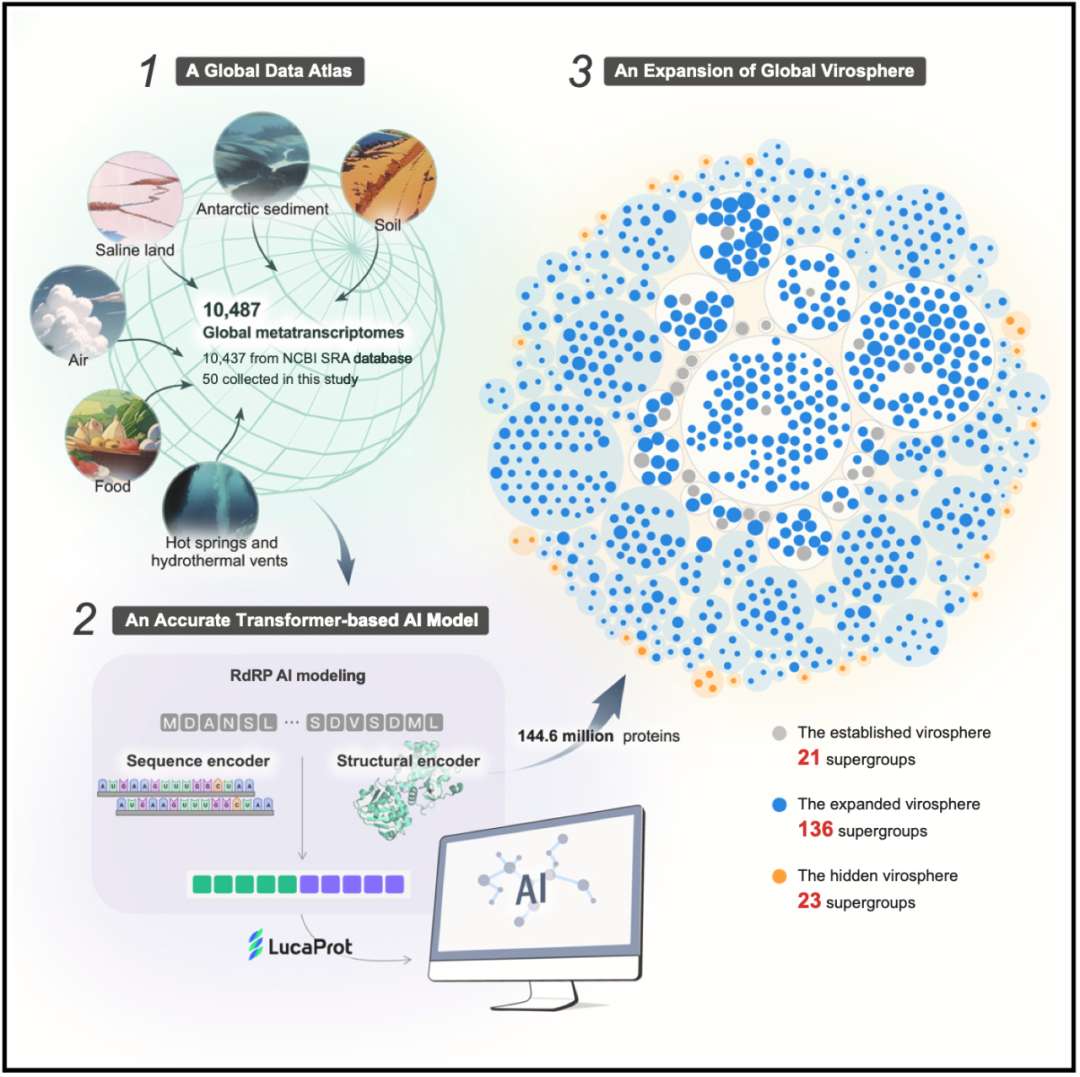
使用AI对全球病毒圈的深度挖掘
总之,该研究基于深度学习在病毒发现领域取得了显著成功,标志着病毒发现新纪元的开启。它不仅拓宽了我们对全球RNA病毒多样性的理解,为解析这些微生物在生态系统中的作用提供新视角。同时,这也为公共卫生、生物安全及疫苗研发等领域带来了启示,有助于提升人类应对未来疫情风险的能力。随着更高效的序列识别技术和快速蛋白质结构预测模型的问世,我们期待,病毒学界将能更高效地运用这些深度学习模型,实现更大规模的病毒发现,并进一步提高识别精确性。
中山大学侯新博士、阿里云贺勇为论文共同第一作者。中山大学医学院施莽教授、阿里云李兆融、悉尼大学Edward Holmes教授为论文通讯作者。
版权声明 本网站所有注明“来源:100医药网”或“来源:bioon”的文字、图片和音视频资料,版权均属于100医药网网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:100医药网”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。 87%用户都在用100医药网APP 随时阅读、评论、分享交流 请扫描二维码下载-> 医药网新闻
医药网新闻
- 相关报道
-
- 登上Cancer Cell:恒瑞医药TROP-2 ADC药物,治疗晚期/转移性实体瘤的首次人试验数据发布 (2025-10-26)
- Science:肾脏应激源引起的线粒体DNA突变可能有助于预测未来的器官衰退 (2025-10-25)
- Nature Genetics:告别拷贝数“估算”时代——ctyper如何利用泛基因组破译重复基因的序列天书? (2025-10-25)
- 《神经元》:科学家首次发现,TREM2-T96K突变与女性患阿尔茨海默病风险增加相关! (2025-10-25)
- 2025年药物警戒任务会议召开 (2025-10-24)
- Nat Commun:皮肤细胞“变身”卵子?科学家开创不孕治疗新路径 (2025-10-24)
- 3D微纳机器人研究获进展 (2025-10-24)
- Science:1+1>2的智慧!蚂蚁的“建筑免疫”如何与个体隔离行为协同增强群体抗性 (2025-10-24)
- 西藏社保系统完成跨越式倒退 织密高原“平易近生保证网” (2025-10-23)
- 重磅!杭州医保新政宣布!来岁1月1日起施行 (2025-10-23)
- 视频新闻
-
- 图片新闻
-
医药网免责声明:
- 本公司对医药网上刊登之所有信息不声明或保证其内容之正确性或可靠性;您于此接受并承认信赖任何信息所生之风险应自行承担。本公司,有权但无此义务,改善或更正所刊登信息任何部分之错误或疏失。
- 凡本网注明"来源:XXX(非医药网)"的作品,均转载自其它媒体,转载目的在于传递更多信息,并不代表本网赞同其观点和对其真实性负责。本网转载其他媒体之稿件,意在为公众提供免费服务。如稿件版权单位或个人不想在本网发布,可与本网联系,本网视情况可立即将其撤除。联系QQ:896150040





















