AlphaFold欢迎对手?Meta-AI预测了超过6亿个蛋白质结构 只用了2周时间 |
 |
来源:生物世界2022-11-04 17:45
但是首尔国立大学的计算生物学家Martin Steinegger表示,他和他的合作者已经使用AlphaFold预测了大约3000万个宏基因组蛋白质的结构。
今年7月,开发AlphaFold的公司DeepMind宣布,将预测的蛋白质结构数据从100万个增加到2.2亿个,而且它不仅关注人类蛋白质,还包括包括植物、细菌、动物和其他生物在内的各种物种的蛋白质结构。它也改变了DNA数据库中几乎所有已知的蛋白质。
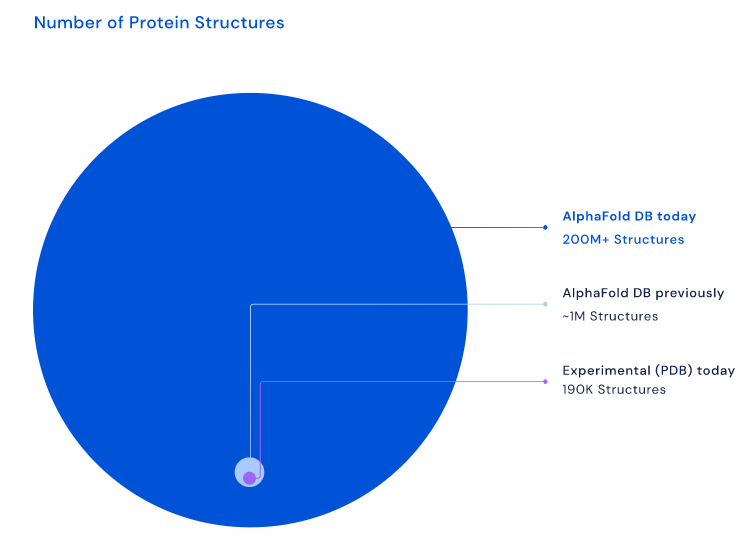
蛋白质结构数量(紫色小圆圈表示实验获得的蛋白质结构数据量,浅蓝色中间圆圈表示早期AlphaFold数据库的数据量,蓝色大圆圈表示当前AlphaFold数据库的数据量)图片来源:deepmind.com
现在,另一个科技巨头Meta(前脸书)正在填补蛋白质宇宙中的暗物质。
Meta的研究人员利用人工智能(AI)预测了超过6亿种蛋白质的结构。这些蛋白质来自细菌、病毒和其他不明微生物。
ESM宏基因组图谱数据库包含6 . 17亿个蛋白质的结构预测。
Meta AI蛋白质团队使用大型语言模型生成了这些结构预测,并在11月1日的预印本上发表了一篇论文[1],描述了这一成果。

Meta AI蛋白质团队的研究负责人亚历山大里维斯(Alexander Rives)表示,这些来自土壤、海洋和人体的微生物中的蛋白质是最不为人知的结构,这些蛋白质非常神秘,可以为我们深入了解生物学提供潜力。
大规模语言模型是一种人工智能(AI)模型,可以从几个字母或单词中预测文本。通常,语言模型是在大量文本的基础上进行训练的。为了将其应用于蛋白质结构的预测,研究小组对已知的蛋白质序列进行了测序。这些蛋白质由20种不同的氨基酸代表,每种氨基酸由一个字母代表。然后,模型学习在氨基酸比例模糊的情况下自动完成蛋白质序列。
蛋白质序列的自动完成
亚历山大里维斯(Alexander Rives)表示,这种训练使模型对蛋白质序列有了直观的认识,而蛋白质序列包含了蛋白质的结构和形状的信息。受DeepMind首创的蛋白质结构工具AlphaFold的启发,这种洞察力与蛋白质结构和序列之间已知关系的信息相结合,从蛋白质序列中生成预测结构。然后,模型学会了在氨基酸比例模糊的情况下自动完成蛋白质。
Meta的研究团队在今年夏天发布的一份报告中表示,其蛋白质结构预测工具ESMFold不如AlphaFold准确,但速度快了约60倍,这意味着结构预测可以扩展到更大的数据库。
作为测试案例,他们决定将这一预测模型应用于一个大型序列DNA数据库的宏基因组,该数据库来自环境,包括土壤、海水、人类肠道、皮肤和其他微生物栖息地。大多数编码潜在蛋白质的DNA序列来自从未培养过的生物,也不为科学界所知。
Meta的团队预测了超过6.17亿个蛋白质的结构。这项工作只花了两周时间。
Alexander Rives表示,这个预测模型是免费的,任何人都可以使用,就像模型的底层代码一样。
在6.17亿个预测的蛋白质结构中,模型认为超过三分之一的预测是高质量的,因此研究人员可以确定蛋白质的整体结构是正确的,在某些情况下,他们可以识别更精细的原子级细节。这些结构中有许多是全新的,不同于通过实验确定的蛋白质结构数据库或从已知生物体预测的AlphaFold数据库中的任何东西。
首尔国立大学的计算生物学家马丁施泰因格(Martin Steinegger)表示,AlphaFold数据库的很大一部分是由彼此几乎相同的结构组成的,而宏基因组数据库应该涵盖了以前从未见过的蛋白质宇宙的很大一部分。这是在黑暗中发现更多蛋白质的绝佳机会。
然而,哈佛大学的进化生物学家谢尔盖奥夫钦尼科夫对ESMFold的上亿个预测表示怀疑,其中一些可能缺乏确定的结构,而另一些可能是非编码DNA,但却被误认为编码蛋白质。似乎还有超过一半的蛋白质空间是我们不知道的。
慕尼黑工业大学的德国计算生物学家布尔夏德罗斯特对ESMFold预测的速度和准确性印象深刻。然而,他也怀疑在从宏基因组数据库预测蛋白质时,它是否比AlphaFold更准确。基于语言模型的预测方法更适合快速确定突变如何改变蛋白质的结构,这是AlphaFold做不到的。他说,我们将看到结构预测变得更加精简、简单和便宜,这将为新事物打开大门。
DeepMind 的一位代表说,该公司目前没有计划在其数据库中包含宏基因组的结构预测,但不排除在未来这样做的可能性。但首尔国立大学的计算生物学家Martin Steinegger表示,他和合作者已经使用AlphaFold来预测大约3000万个宏基因组蛋白质的结构。他们希望从中发现新的RNA病毒种类。他认为,这类预测工具的下一步显然是研究生物学中的暗物质。预计我们将很快会在这些宏基因组结构的分析方面出现爆炸式增长。
版权声明 本网站所有注明“来源:100医药网”或“来源:bioon”的文字、图片和音视频资料,版权均属于100医药网网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:100医药网”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。 87%用户都在用100医药网APP 随时阅读、评论、分享交流 请扫描二维码下载-> 医药网新闻
医药网新闻
- 相关报道
-
- Nat Biomed Eng:用“声波画笔”调控大脑!新型全息超声技术有望打开人类神经疾病治疗新大门 (2025-10-07)
- 《神经元》:短期高脂饮食也同样危险!科学家发现,两天的高脂饮食即可激活小鼠大脑特定抑制性神经元,导致认知受损,恢复正常饮食可逆转 (2025-10-07)
- 相分离研究登上Nature:超越转录调控,mRNA协调易凝聚蛋白稳态 (2025-10-07)
- 动辄百万美元奖金,这些科学大奖想要与诺贝尔奖竞争:它们含金量如何? (2025-10-06)
- Nature:T细胞耗竭的悖论——并非“精疲力竭”,而是“生产过剩”引发的蛋白毒性风暴 (2025-10-06)
- Cell:沉默的基因组,喧嚣的转录场——解密结核菌适应性的隐藏驱动力 (2025-10-06)
- Nature:谁掌控了人类生命的第一个开关?一个被“驯化”的逆转录病毒的自白 (2025-10-06)
- 三个月,改写生命!全球首例帕金森病实现功能性治愈 (2025-10-05)
- AJP:迄今最长时间真实世界安全性分析显示,艾司氯胺酮鼻喷剂的安全性与临床研究和产品说明书中已确定的一致 (2025-10-05)
- Nature子刊:谭蔚泓/邱丽萍团队开发核酸适配体武装的单核细胞疗法,治疗阿尔茨海默病 (2025-10-05)
- 视频新闻
-
- 图片新闻
-
医药网免责声明:
- 本公司对医药网上刊登之所有信息不声明或保证其内容之正确性或可靠性;您于此接受并承认信赖任何信息所生之风险应自行承担。本公司,有权但无此义务,改善或更正所刊登信息任何部分之错误或疏失。
- 凡本网注明"来源:XXX(非医药网)"的作品,均转载自其它媒体,转载目的在于传递更多信息,并不代表本网赞同其观点和对其真实性负责。本网转载其他媒体之稿件,意在为公众提供免费服务。如稿件版权单位或个人不想在本网发布,可与本网联系,本网视情况可立即将其撤除。联系QQ:896150040





















