基因编辑领域最新研究进展 |
 |
在此之前,美国哈佛大学的David R. Liu和他的同事们开发出一种新的称为引导编辑(prime editing)的基因组编辑方法。这种方法使用工程化的Cas9切口酶(H840A)-逆转录酶(RT)融合蛋白和引导编辑向导RNA(prime editing guide RNA, pegRNA),可在人细胞中进行所需的编辑。
在一项新的研究中,中国科学院遗传与发育生物学研究所的高彩霞(Gao Caixia)教授及其研究团队对一种引导编辑系统(prime editing system, PPE)进行优化,从而在两种主要的谷类作物中产生所需的点突变、 插入和缺失。PPE系统的主要成分是Cas9切口酶-RT融合蛋白和pegRNA。相关研究 结果近期发表在Nature Biotechnology期刊上,论文标题为“Prime genome editing in rice and wheat”。
通过使用这种PPE系统,这些研究人员在原生质体中在9个水稻位点和7个小麦位点上产生了所有12种类型的单碱基替换,以及多种点突变和小DNA片段插入,效率高达19.2%。这种PPE系统的编辑效率受到引导结合位点(primer binding site, PBS)和RT模板长度的强烈影响。
尽管这种PPE系统会产生副产物(脱靶效应),但是可以通过优化RT模板长度来减少这些副产物。此外,通过使用针对植物优化的PPE系统,他们发现初始的RT可以被CaMV-RT(来自花椰菜花叶病毒)和反转录子衍生性RT(来自大肠杆菌BL21)替换。通过使用PPE-Ribozyme(PPE-R)并在37°C下孵育,针对一些靶标的引导编辑效率也可得到改善。
此外,高彩霞教授和她的合作者能够构建稳定的携带G-to-T点突变、多核苷酸替换和许多所需的6nt缺失的突变水稻植株,它们的产生效率接近22%。值得注意的是,使用当前的编辑工具很难产生这三种类型的突变。
高彩霞教授说,“尽管这种PPE系统的效率低于碱基编辑器,但是它仍然是一种吸引人的新工具,可用于产生所有12种类型的单点突变、不同替换的混合物以及插入和缺失。这种系统因此具有巨大的潜力用于开展植物育种和功能基因组学研究。”
DOI:10.1038/s41589-020-0490-4
“基因魔剪”CRISPR-Cas9引起能在特殊靶向位点切割DNA而彻底改变了遗传学领域的研究,如今研究人员能利用Cas9酶来专门关闭基因的表达,或将新型DNA片段插入到基因组中,但不论Cas9有多么专一特殊,其都会切开一些不该切开的位点;近日,一项刊登在国际杂志Nature Chemical Biology上的研究报告中,来自德国马丁路德大学的科学家们通过研究报道了一种新型的Cas9突变体,其或能增加基因编辑的特异性。
为了能让Cas9切割DNA靶点,其就需要在导向RNA的带领下被引入到靶点位置,导向RNA含有DNA靶点的互补序列,其工作原理就好像是邮政编码一样能将Cas9引导到目的靶点,然而有时候Cas9会切割与实际靶点非常相似的DNA序列,这就被称为脱靶效应;CRISPR-Cas9不受欢迎的特性就是可能会导致基因编辑的不准确性,在人类基因组中的错误位置出现意外的切割可能会产生非常深远的影响。
如今科学家们尝试利用不同的方法来优化Cas9的特异性,当前研究中,研究人员就对Cas9中名为螺旋桥(bridge helix)的进化保守结构域进行了深入研究。研究者发现,这种螺旋桥在Cas9与其导向RNA和DNA靶点相互作用上的机制上扮演着关键角色,随后他们识别出了一组氨基酸残基,其能与导向RNA的磷酸骨架接触,从而促进稳定回路结构的形成,后者对于Cas9的活性非常重要,在这种回路结构中,Cas9结合导向RNA就能与DNA靶向序列的互补链进行配对,同时还会替换第二股DNA链,从而使得Cas9就能切割两条DNA链。
研究者通过改变这些氨基酸残基就能产生新的Cas9突变体,他们发现,相比原始的Cas9酶而言,多个突变体切割脱靶位点的频率明显变低了,后期深入研究后研究者发现,其中一种名为R63A/Q768A的突变体还能够增加人类细胞中Cas9基因编辑的特异性;本文研究结果或为后期科学家们有效优化CRISPR-Cas9奠定了基础,目前研究人员还需要后期进行更为深入的研究来解析CRISPR-Cas系统的生化特性从而有效改善其编辑准确性。
doi:10.1126/sciadv.aax2941.
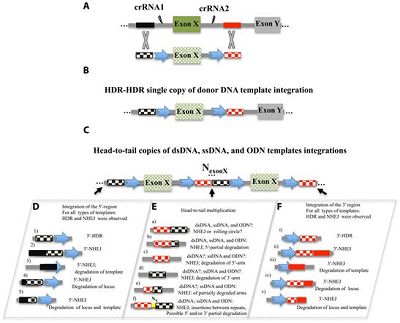
在一项新的研究中,来自德国明斯特大学的研究人员发现,在小鼠进行常规的CRISPR-Cas9基因插入过程中,不必要的DNA重复频率很高。相关研究结果发表在2020年2月21日的Science Advances期刊上,论文标题为“Pervasive head-to-tail insertions of DNA templates mask desired CRISPR-Cas9–mediated genome editing events”。他们描述了他们如何发现不必要的DNA重复,并针对这一点提醒了其他的研究人员。
医药网新闻
- 相关报道
-
- Nature Methods:蛋白质“变形金刚”设计指南——AI“脑补”自然进化,一键生成智能分子开关 (2025-08-10)
- N Engl J Med:改写移植规则!从“免疫耐受”迈向“免疫规避”的新纪元 (2025-08-10)
- Science重磅:破解阿尔茨海默病抗体药的脑出血副作用——突破血脑屏障,靶向递送抗Aβ抗体 (2025-08-10)
- Nat Rev Neurosci重要综述:自主神经系统的分子和功能多样性——系统回顾了该领域近十年来的关键进展 (2025-08-09)
- Cancer Cell:于君团队证实,补充这种益生菌,可增强结直肠癌免疫治疗效果 (2025-08-09)
- Cancer Cell:魏嘉/李颜团队揭示未成熟中性粒细胞在癌症骨转移中的关键作用,并提出治疗新策略 (2025-08-09)
- 为何CAR-T 疗法易复发?Mol Cancer:细胞衰老成“隐形杀手”,共刺激域差异决定疗效命运 (2025-08-08)
- Nature:多发性硬化症的“元凶”现身?科学家揭示EB病毒惊人发现! (2025-08-08)
- Nature:锂缺乏或会诱发阿尔兹海默病,但锂化合物却能逆转小鼠疾病 (2025-08-08)
- 不止供能,还能抗癌?!Cell Metabol:葡萄糖能帮抗癌 T 细胞“充电”!糖鞘脂合成是关键 (2025-08-08)
- 视频新闻
-
- 图片新闻
-
医药网免责声明:
- 本公司对医药网上刊登之所有信息不声明或保证其内容之正确性或可靠性;您于此接受并承认信赖任何信息所生之风险应自行承担。本公司,有权但无此义务,改善或更正所刊登信息任何部分之错误或疏失。
- 凡本网注明"来源:XXX(非医药网)"的作品,均转载自其它媒体,转载目的在于传递更多信息,并不代表本网赞同其观点和对其真实性负责。本网转载其他媒体之稿件,意在为公众提供免费服务。如稿件版权单位或个人不想在本网发布,可与本网联系,本网视情况可立即将其撤除。联系QQ:896150040






















