Cell Metabolism:上海交大陈豪燕团队等揭示肠道菌群及其代谢物对癌症免疫治疗的影响 |
 |
来源:生物世界 2025-02-06 13:46
这项研究揭示了肠道微生物组、肠道代谢组与免疫治疗反应之间的相互作用,确定了可能的生物标志物以改善治疗效果。上海交通大学医学院附属仁济医院陈豪燕、肖秀英,苏州大学第一附属医院何宋兵、江苏省人民医院沈波等人在Cell 子刊CellMetabolism 上发表了题为:Interplay between gut microbial communities and metabolites modulates pan-cancer immunotherapy responses 的研究论文。
该研究利用多组学分析,揭示了肠道微生物群和其代谢产物之间的相互作用调节泛癌症治疗应答。

在这项最新研究中,研究团队对 165 名接受抗 PD-1/ PD-L1 治疗的患者的粪便微生物组和代谢组进行了多组学分析,确定了与治疗反应相关的微生物和代谢物。
研究团队通过对来自四个公共宏基因组数据集(n = 568)的数据进行整合,发现了跨队列的微生物和代谢特征,并在一个独立队列(n = 138)中得到了验证。一个融合了这些特征的综合预测模型表现出了强大的性能。
值得注意的是,该研究确定了五种与治疗反应相关的肠型,每种肠型都与特定的细菌分类群和代谢物相关联。
在这些代谢物中,苯乙酰谷氨酰胺(PAGln)与治疗反应呈负相关,并且在小鼠体内实验中被证明会削弱抗 PD-1 单抗的疗效。
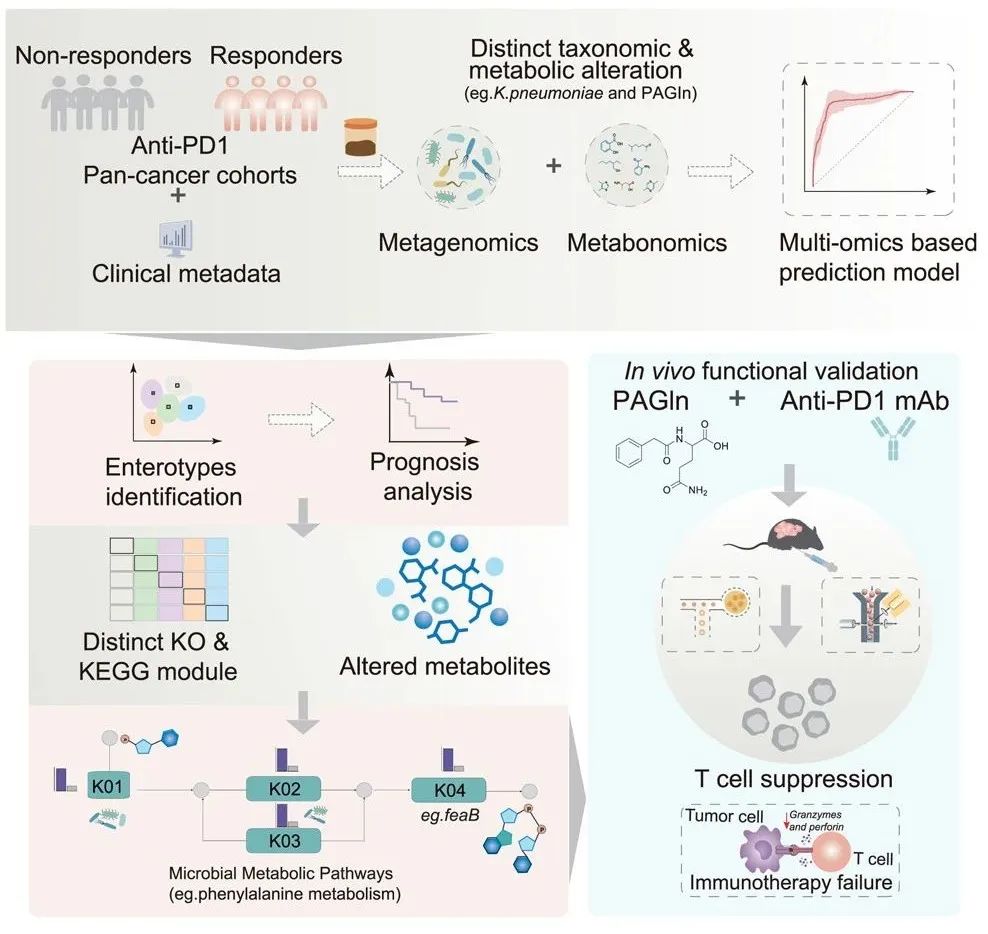
该研究的核心发现:
多组学揭示了微生物组与代谢组之间的联系,这对免疫检查点阻断疗法的反应至关重要;
在多个队列中验证来预测性微生物和代谢特征;
五种具有独特代谢特征的肠型突显了其在免疫检查点阻断治疗反应中的作用;
PAGln 是一种微生物代谢产物,被确认为降低抗 PD-1 单抗疗效的关键因素。
总的来说,这项研究揭示了肠道微生物组、肠道代谢组与免疫治疗反应之间的相互作用,确定了可能的生物标志物以改善治疗效果。 版权声明 本网站所有注明“来源:100医药网”或“来源:bioon”的文字、图片和音视频资料,版权均属于100医药网网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:100医药网”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。 87%用户都在用100医药网APP 随时阅读、评论、分享交流 请扫描二维码下载-> 医药网新闻
医药网新闻
- 相关报道
-
- 11家创新药公司完成新一轮融资! (2025-09-25)
- 自闭症社交障碍有破解之法?Nat Commun:大脑里的“社交激素”通路断了,激活受体就能修复 (2025-09-25)
- 不分时间“秒睡”,警惕这种被忽视的病!全球数十款新药加速冲刺,正在为患者点亮希望 (2025-09-25)
- 每月吃 1 片就能防 HIV ?PLoS Biol:一种新型HIV抗逆转录病毒口服药来了,长效还抗耐药 (2025-09-25)
- 益生菌来源 EVs 攻克脂肪肝!Front Immunol:无需活菌定植的“益生菌精华”,降肝酶、稳血脂还护肠道 (2025-09-25)
- 《自然》子刊:鼻腔菌群也能影响大脑!上海交大/上科大团队发现,鼻腔金黄色葡萄球菌通过降解性激素介导抑郁行为 (2025-09-25)
- 老药新用!黄波团队发现碳酸锂可逆转肺纤维化,清除“瘢痕制造者”肌成纤维细胞 (2025-09-25)
- Cell:一种保护性涂层有望让通用型CAR-T细胞疗法成为可能 (2025-09-25)
- 原来植物也有“疗伤因子”!ACS Appl Mater Interfaces:丹参来源囊泡,让受损神经在大鼠身上“重连” (2025-09-25)
- 精鼎医药宣布任命 Rob Goodwin 担任公司首席运营官 (2025-09-24)
- 视频新闻
-
- 图片新闻
-
医药网免责声明:
- 本公司对医药网上刊登之所有信息不声明或保证其内容之正确性或可靠性;您于此接受并承认信赖任何信息所生之风险应自行承担。本公司,有权但无此义务,改善或更正所刊登信息任何部分之错误或疏失。
- 凡本网注明"来源:XXX(非医药网)"的作品,均转载自其它媒体,转载目的在于传递更多信息,并不代表本网赞同其观点和对其真实性负责。本网转载其他媒体之稿件,意在为公众提供免费服务。如稿件版权单位或个人不想在本网发布,可与本网联系,本网视情况可立即将其撤除。联系QQ:896150040





















