Cell:郭国骥团队开发女娲系列模型破译脊椎动物基因组天书 |
 |
来源:BioArt 2025-07-14 10:12
这项研究不仅开发了一款超高通量超高灵敏度的细胞图谱绘制工具,更创造了强大的基因组AI模型。脊椎动物基因组的调控序列语法仍未被完全解析。为此浙江大学医学院/良渚实验室郭国骥教授团队郭国骥组Cell报道首个哺乳动物细胞图谱和首个人类细胞图谱。在细胞图谱数据的基础上,团队建立了基因组AI模型Nvwa(女娲),首次实现了完全基于基因组序列预测单细胞分辨率下的基因表达;开发了基因组突变效应预测模型Huatuo(华佗),首次实现人体全身细胞类型特异性表观修饰(2000多种表观特征)和基因表达量的预测,并构建了44种主要细胞类型的特异性基因调控遗传变异景观(Nature Communications,2023)。为了进一步提升基因组AI模型的精准度和泛化能力,团队从生命表型的测量技术和模型训练的数据质量入手开展了持续性的研究。
2025年7月8日,浙江大学医学院/良渚实验室郭国骥教授团队在Cell期刊上发表了题为Modeling the vertebrate regulatory sequence landscape by UUATAC-seq and deep learning的研究论文。在这项工作中郭国骥团队建立了超高通量超灵敏单核ATAC测序技术(UUATAC-seq),可在单日内高效率高质量的完成一个物种的染色质可及性图谱。基于该技术,团队为五大代表性脊椎动物中绘制候选顺式调控元件图谱,开发多任务深度学习模型女娲CE,并实现了从基因组序列到单细胞水平调控元件图谱的直接预测。团队发现,脊椎动物调控语法的保守性明显强于核苷酸序列本身,且该语法将脊椎动物调控原件序列在高维分类为不同的功能模块,由此揭示细胞类型特异性基因表达的序列基础。另外,女娲CE模型在多项指标上,超越现有的基因组AI模型,并能精准预测合成突变对谱系特异性调控元件功能的影响。最后,团队利用基因编辑实验,首次验证了完全由人工智能设计的人类疾病治愈性位点。这项工作为全面解读基因组语言和建立数字生命模型奠定了坚实的基础。

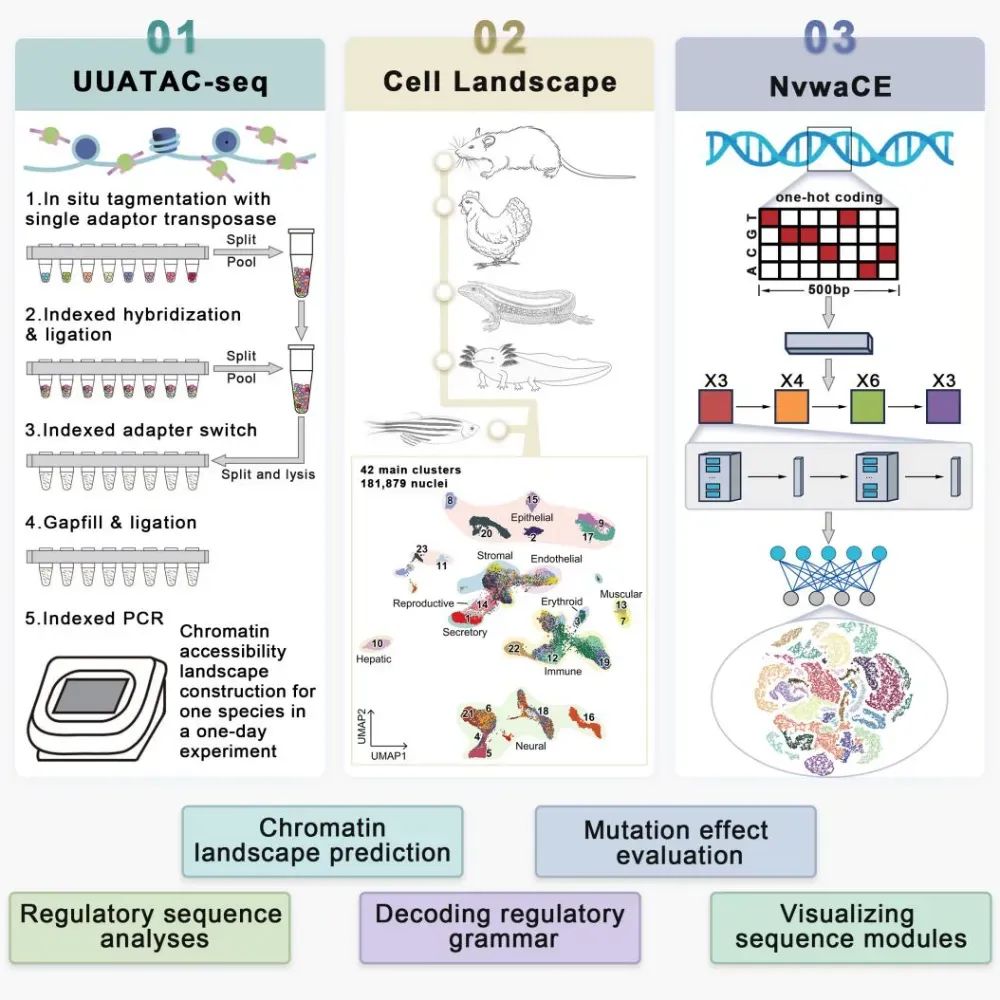
该工作自主研发了超高通量、超高灵敏度单细胞测序平台UUATAC-seq(ultra-throughput ultra-sensitive single-nucleus ATAC-seq)。UUATAC-seq实现了技术上的双重突破:创新性的双端同型转座酶切设计结合体外精准温度控制策略,使其达到了前所未有的超高灵敏度;同时,独特的四轮组合标签策略赋予其超高通量的能力,显著超越了现有其它单细胞测序技术。UUATAC-seq一次实验获得的有用信息与现有技术相比实现了数量级的提升,平台的强大优势为其广泛应用奠定了坚实基础。该技术革新性地支持在染色质开放区域以DNA双链同时为模板进行测序(双链模板测序),这一关键优势成功突破了过往单链测序技术(如10X ATAC)的局限,极大地拓展了对开放染色质区域的解读维度。同时,UUATAC-seq展现出卓越的样本兼容性,完美支持对新鲜样本以及经固定处理的样本进行高效测序分析。这一显著优势彻底解决了传统方法因过分依赖固定样本而导致的TSS(转录起始位点)偏好性难题,大幅提升了染色质开放区域检测的准确性和可靠性,为更多样化的研究场景提供了有力工具。
依托这一革命性技术,研究团队对涵盖五大脊椎动物纲目的代表性物种(哺乳类-小鼠、鸟类-鸡、爬行类-守宫、两栖类-蝾螈、水生类-斑马鱼)进行了系统性分析,成功构建了跨物种的高质量全身染色质可及性图谱,并基于对数百万顺式调控元件的深度解析,揭示了脊椎动物细胞类型演化中保守与特异性调控程序的精妙机制。研究发现,基因组大小与开放染色质区域的数量高度相关,而单个开放区域的大小在不同脊椎动物间表现出一致性。
为进一步解析隐藏在海量调控元件背后的复杂 语法 ,该研究提出一个深度学习模型女娲CE,能够基于脊椎动物的基因组序列,在单细胞分辨率上预测其在任意脊椎动物细胞类型中的染色质可及性水平。值得一提的是,女娲CE凭借泛化能力能够从基因组序列出发预测未经训练物种的染色质可及性蓝图,并一次性预测了包括人、猴、牛、猪、马、羊、熊猫七个物种的单细胞调控原件蓝图。其种,对于人类调控元件可及性水平的预测值与实验测量值显示出较好的相关性。此外,模型能够准确地预测基因组非编码区突变的调控效应,在人类饱和突变数据集上,其预测非编码区突变调控效应的准确性显著优于其他基因组AI模型。功能实验验证表明,经女娲CE预测出的一个镰刀型贫血症治愈性基因位点(HBG1-68:A G)在经过基因编辑后,能实现胎儿血红蛋白表达量的显著提升。这一结果首次在人体细胞中证明了基因组AI模型精准预测功能性位点的性能。
与Deepmind团队预发表的Alphagenome模型相比,浙江大学的女娲系列模型不依赖ENCODE的复杂数据体系,能够实现单细胞水平的序列功能预测,数据集包涵更多的在体细胞类型。与此同时,斯坦福大学与英伟达研究院预发表的evo2模型,主要擅长分析的是单细胞生物,且尚不具备理解脊椎动物细胞类型的能力。女娲系列模型表现出了强大的泛化能力,并能实现跨物种基因组到单细胞水平调控元件蓝图的直接预测。女娲CE模型虽然放弃了长DNA序列的输入模式,但其运用的分段式扫描策略节省了大量计算资源,并为后续调控原件相互作用的建模奠定了基础。另最后女娲CE模型的建立,基于迄今为止最高质量的单细胞图谱数据,并对几乎所有的细胞类型实现了AUROC 0.90的预测准确率,这是其他基因组AI模型无法企及的。
综上,这项研究不仅开发了一款超高通量超高灵敏度的细胞图谱绘制工具,更创造了强大的基因组AI模型。首先,该研究将赋能合成生物学,模型的预测结果可用于设计具备特定调控功能的合成DNA,并实现特定的表型;其次,该研究将加深人类对遗传疾病的理解,并预测全新的治愈性位点;最后,该研究将能辅助农业育种,设计更高产的畜牧业物种基因组。该研究范式将对生命科学,医学和农学研究带来深远影响。
华东师范大学李大力团队、北京师范大学王晓群团队、浙江大学伍赛团队、贺诗波团队提供了大力帮助。浙江大学医学院公共技术平台及良渚实验室公共技术平台提供了支撑。
原文链接:
https://doi.org/10.1016/j.cell.2025.06.020
: , , , , , , , , , , , , 。 视频 小程序 赞 ,轻点两下取消赞 在看 ,轻点两下取消在看 分享 留言 收藏 听过
版权声明 本网站所有注明“来源:100医药网”或“来源:bioon”的文字、图片和音视频资料,版权均属于100医药网网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:100医药网”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。 87%用户都在用100医药网APP 随时阅读、评论、分享交流 请扫描二维码下载-> 医药网新闻
医药网新闻
- 相关报道
-
- 动辄百万美元奖金,这些科学大奖想要与诺贝尔奖竞争:它们含金量如何? (2025-10-06)
- Nature:T细胞耗竭的悖论——并非“精疲力竭”,而是“生产过剩”引发的蛋白毒性风暴 (2025-10-06)
- Cell:沉默的基因组,喧嚣的转录场——解密结核菌适应性的隐藏驱动力 (2025-10-06)
- Nature:谁掌控了人类生命的第一个开关?一个被“驯化”的逆转录病毒的自白 (2025-10-06)
- 三个月,改写生命!全球首例帕金森病实现功能性治愈 (2025-10-05)
- AJP:迄今最长时间真实世界安全性分析显示,艾司氯胺酮鼻喷剂的安全性与临床研究和产品说明书中已确定的一致 (2025-10-05)
- Nature子刊:谭蔚泓/邱丽萍团队开发核酸适配体武装的单核细胞疗法,治疗阿尔茨海默病 (2025-10-05)
- 多学科合作精准拆弹!仁济医院成功救治白塞病合并胸主动脉瘤破裂出血患者 (2025-10-05)
- Nature:自闭症的“双面人生”——迟来的诊断背后,是另一套基因密码吗? (2025-10-05)
- 中国科学院最新论文登上Cell头条 (2025-10-04)
- 视频新闻
-
- 图片新闻
-
医药网免责声明:
- 本公司对医药网上刊登之所有信息不声明或保证其内容之正确性或可靠性;您于此接受并承认信赖任何信息所生之风险应自行承担。本公司,有权但无此义务,改善或更正所刊登信息任何部分之错误或疏失。
- 凡本网注明"来源:XXX(非医药网)"的作品,均转载自其它媒体,转载目的在于传递更多信息,并不代表本网赞同其观点和对其真实性负责。本网转载其他媒体之稿件,意在为公众提供免费服务。如稿件版权单位或个人不想在本网发布,可与本网联系,本网视情况可立即将其撤除。联系QQ:896150040





















